2023-11-20
来源:上海百沐生物科技有限公司
所属分类:单细胞测序100问
在 scRNA-seq 数据分析中,我们通过查找与已知细胞状态或细胞周期阶段相关的细胞身份来描述数据集中的细胞结构。这个过程通常称为细胞身份注释。为此,我们将细胞组织成簇,以推断相似细胞的身份。聚类本身是一个常见的无监督机器学习问题。我们可以通过最小化缩减表达空间中的簇内距离来导出簇。在这种情况下,表达空间决定了细胞相对于降维表示的基因表达相似性。例如,这种较低维度的表示是通过主成分分析确定的,然后基于欧几里德距离进行相似性评分。
在 KNN 图中,节点由反映数据集中的单元格组成。我们首先在 PC 缩减表达空间上计算所有细胞的欧几里德距离矩阵,然后将每个细胞连接到其 K 个最相似的细胞。通常,K 设置为 5 到 100 之间的值,具体取决于数据集的大小。KNN 图通过将表达空间的密集区域表示为图中的密集连接区域来反映表达数据的基础拓扑[ Wolf et al. ,2019 ]。KNN 图中的密集区域是通过 Leiden 和 Louvain 等社区检测方法检测的[ Blondel等人。,2008 ]。
Leiden算法是Louvain算法的改进版本,在单细胞RNA-seq数据分析方面优于其他聚类方法([ Du et al. , 2018 , Freytag et al. , 2018 , Weber and Robinson, 2016 ])。由于 Louvain 算法不再维护,因此首选使用 Leiden。
因此,我们建议使用 Leiden 算法[ Traag等人。,2019 ]在单细胞 k 最近邻(KNN)图上对单细胞数据集进行聚类。莱顿通过考虑簇中单元之间的链接数量与数据集中的总体预期链接数量来创建簇。
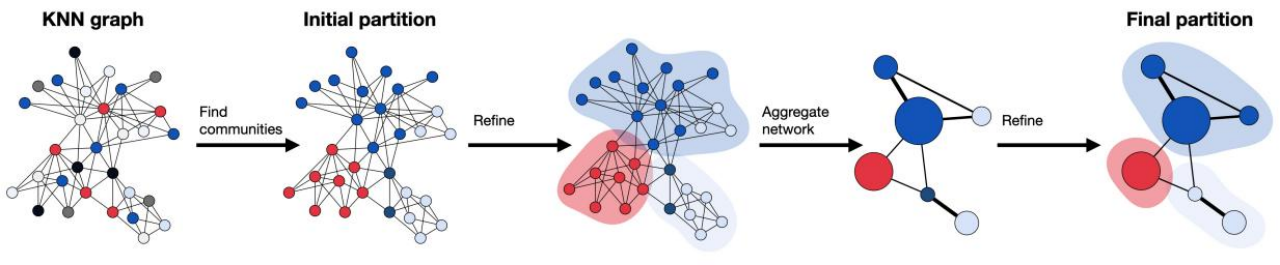
注:Leiden 算法在从 PC 简化表达空间获得的 KNN 图上计算聚类。它从一个初始分区开始,其中每个节点都来自自己的社区。接下来,算法将单个节点从一个社区移动到另一个社区以找到一个分区,然后对其进行细化。基于细化的分区生成聚合网络,再次细化直至无法获得进一步的改进,从而达到最终的分区。
起点是一个单例分区,其中每个节点都充当自己的社区。下一步,该算法通过将单个节点从一个社区移动到另一个社区来创建分区,随后进行细化以增强分区。然后将细化的分区聚合到网络。随后,算法再次移动聚合网络中的各个节点,直到细化不再改变分区。重复所有步骤,直到创建最终的聚类并且分区不再发生变化。
Leiden 模块具有分辨率参数,可以确定分区簇的规模,从而确定聚类的粗糙度。更高分辨率的参数会导致更多的簇。该算法还允许通过对 KNN 图进行子设置来对数据集中的特定聚类进行有效的子聚类。子聚类使用户能够识别聚类内的细胞类型特定状态或更精细的细胞类型标记,但也可能导致仅由于数据中存在的噪声而产生的模式。
 在线咨询
在线咨询 微信咨询
微信咨询

 电话咨询
电话咨询
联系电话:191 2100 2160
 顶部
顶部
百沐生物孵化于复旦大学,核心团队由复旦大学、中科院、协和医院等知名科研院所的卓越科学家以及来自阿里、美团、小米、生物医药集团等头部互联网科技公司和大型药企集团的综合背景精英组成,专注于将人工智能(AI)技术与生物技术(BT)深度融合,为生命科学研究和精准医疗领域带来创新解决方案。 公司核心平台为自研单细胞平台(BMBIOS)和小沐AI平台(AIGOBIO)。

微信联系我们