2023-11-15
来源:上海百沐生物科技有限公司
所属分类:单细胞测序100问
质量控制的第一步是从数据集中删除低质量的读数。当细胞检测到的基因数量较少、计数深度较低且线粒体计数较高时,细胞膜可能会破裂,这表明细胞正在死亡。由于这些细胞通常不是我们分析的主要目标,并且可能会扭曲我们的下游分析,因此我们在质量控制过程中将其去除。为了识别它们,我们定义了细胞质量控制(QC)阈值。细胞质控通常对以下三个质控协变量进行:
每个条形码的计数数量(计数深度)
每个条形码的基因数量
每个条形码的线粒体基因计数分数
在细胞 QC 中,这些协变量通过阈值过滤,因为它们可能对应于死亡细胞。正如所指出的,它们可能反映了膜破裂的细胞,其细胞质 mRNA 已泄漏,因此只有线粒体中的 mRNA 仍然存在。这些细胞可能会显示出较低的计数深度、很少检测到的基因以及较高比例的线粒体读数。然而,共同考虑三个 QC 协变量至关重要,否则可能会导致细胞信号的误解。例如,线粒体计数相对较高的细胞可能参与呼吸过程,不应被过滤掉。然而,计数低或高的细胞可能对应于静止细胞群或尺寸较大的细胞。因此,在对单个协变量做出阈值决策时,优选考虑多个协变量。一般来说,建议排除较少的细胞并尽可能允许,以避免过滤掉活细胞
群或小亚群。
仅对少数或小型数据集进行 QC 通常以手动方式执行,方法是查看不同 QC 协变量的分布并识别随后将被过滤的异常值。然而,随着数据集规模的增长,这项任务变得越来越耗时,可能值得考虑通过 MAD(中值绝对偏差)进行自动阈值处理。
在 QC 中,第一步是计算 QC 协变量或指标。我们使用 scanpy 函数来计算这些 sc.pp.calculate_qc_metrics,该函数还可以计算特定基因群体的计数比例。因此,我们定义了线粒体、核糖体和血红蛋白基因。值得注意的是,线粒体计数用前缀“mt-”或“MT-”注释,具体取决于数据集中考虑的物种。如前所述,本笔记本中使用的数据集是人骨髓,因此线粒体计数用前缀“MT-”注释。对于鼠标数据集,前缀通常是小写,即“mt-”。
# mitochondrial genes
adata.var["mt"] = adata.var_names.str.startswith("MT-")
# ribosomal genes
adata.var["ribo"] = adata.var_names.str.startswith(("RPS", "RPL"))
# hemoglobin genes.
adata.var["hb"] = adata.var_names.str.contains(("^HB[^(P)]"))
我们现在可以使用 scanpy 计算相应的 QC 指标。
sc.pp.calculate_qc_metrics(
adata, qc_vars=["mt", "ribo", "hb"], inplace=True, percent_top=[20], log1p=True
)
adata
AnnData object with n_obs × n_vars = 16934 × 36601
obs:'n_genes_by_counts','log1p_n_genes_by_counts','total_counts','log1p_total_counts','pct_counts_in_top_20_genes','total_counts_mt','log1p_total_counts_mt','pct_counts_mt','total_counts_ribo','log1p_total_counts_ribo','pct_counts_ribo','total_counts_hb', 'log1p_total_counts_hb', 'pct_counts_hb'
正如我们所看到的,该函数向.var和.obs添加了几个附加列。在这里重点介绍其中的一些,有关不同指标的更多信息可以在 scanpy 文档中找到:
n_genes_by_countsin.obs是细胞中计数呈阳性的基因数量。
total_counts是细胞的计数总数,这也可能称为库大小。
pct_counts_mt是线粒体细胞总数的比例。
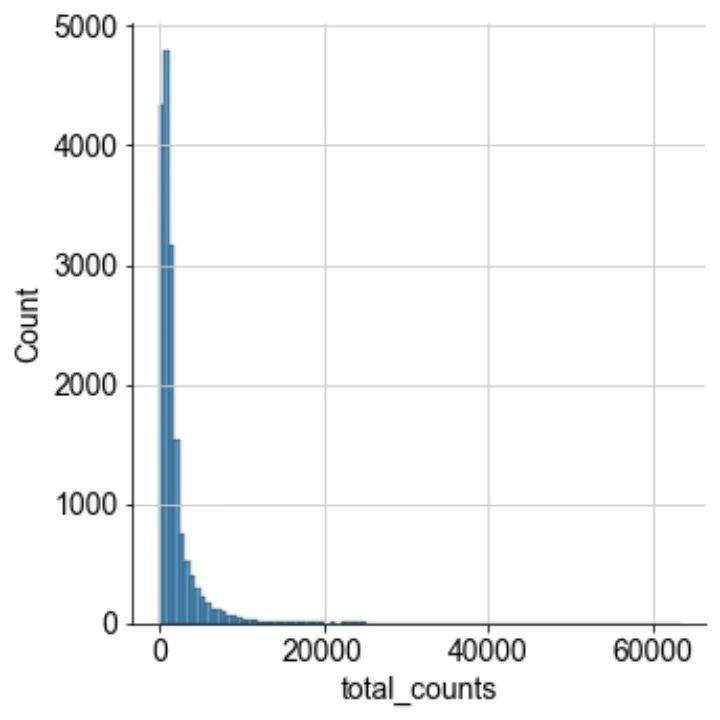
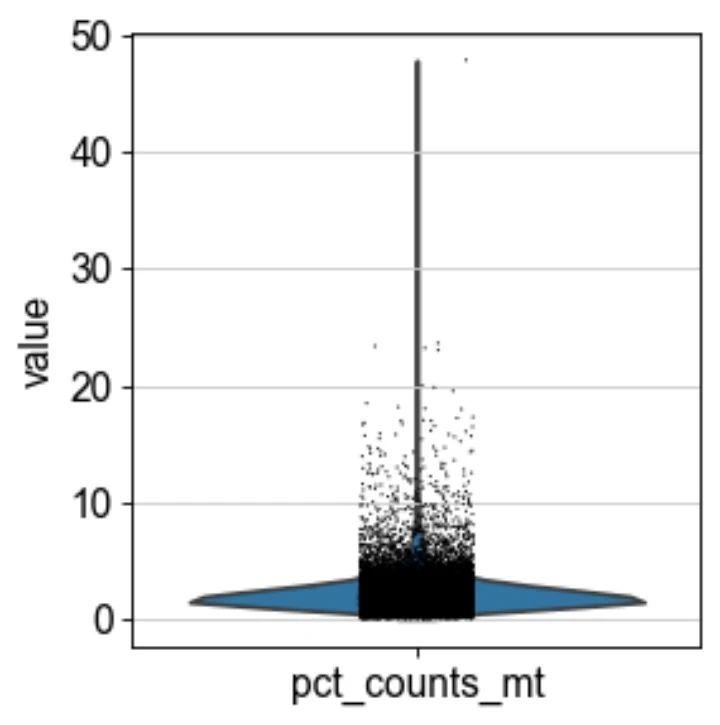
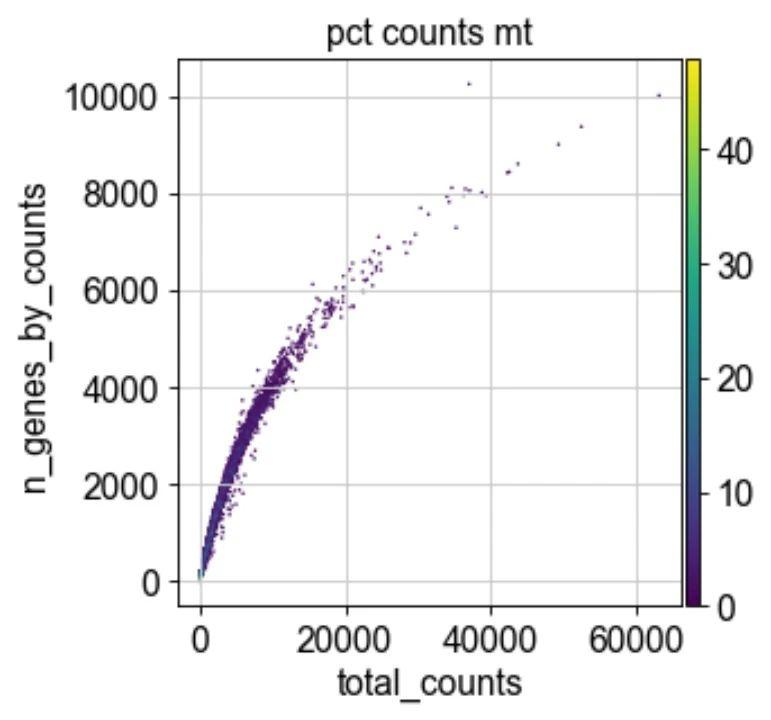
我们现在绘制三个样本的QC协变量 n_genes_by_counts, total_counts and pct_counts_mt,以评估各个细胞的捕获情况。
p1 = sns.displot(adata.obs["total_counts"], bins=100, kde=False)
# sc.pl.violin(adata, 'total_counts')
p2 = sc.pl.violin(adata, "pct_counts_mt")
p3 = sc.pl.scatter(adata, "total_counts", "n_genes_by_counts", color="pct_counts_mt")
... storing 'feature_types' as categorical
... storing 'genome' as categorical
图一
图二
图三
这些图表明,一些读数具有相对较高百分比的线粒体计数,通常与细胞降解相关。但由于每个细胞的计数数量足够高,并且大多数细胞的线粒体读数百分比低于20%,我们仍然可以处理数据。基于这些图,现在还可以定义用于过滤细胞的手动阈值。这里我们将展示基于 MAD的自动阈值和过滤的QC。
首先,我们定义一个函数。
def is_outlier(adata, metric: str, nmads: int):
M = adata.obs[metric]
outlier = (M < np.median(M) - nmads * median_abs_deviation(M)) | (
np.median(M) + nmads * median_abs_deviation(M) < M
)
return outlier
我们现在将此函数应用于log1p_total_counts、log1p_n_genes_by_counts和pct_counts_in_top_20_genesQC协变量,每个协变量的阈值为5 MADs。
adata.obs["outlier"] = (
is_outlier(adata, "log1p_total_counts", 5)
| is_outlier(adata, "log1p_n_genes_by_counts", 5)
| is_outlier(adata, "pct_counts_in_top_20_genes", 5)
)
adata.obs.outlier.value_counts()
False 16065
True 869
Name: outlier, dtype: int64
pct_counts_Mt使用3个MADs进行过滤。此外,线粒体计数百分比超过 8% 的细胞也会被滤除。
adata.obs["mt_outlier"] = is_outlier(adata, "pct_counts_mt", 3) | (
adata.obs["pct_counts_mt"] > 8
)
adata.obs.mt_outlier.value_counts()
False 15240
True 1694
Name: mt_outlier, dtype: int64
现在,我们根据这两个附加列来过滤 AnnData 对象。
print(f"Total number of cells: {adata.n_obs}")
adata = adata[(~adata.obs.outlier) & (~adata.obs.mt_outlier)].copy()
print(f"Number of cells after filtering of low quality cells: {adata.n_obs}")
Total number of cells: 16934
Number of cells after filtering of low quality cells: 14814
p1 = sc.pl.scatter(adata, "total_counts", "n_genes_by_counts", color="pct_counts_mt")
 在线咨询
在线咨询 微信咨询
微信咨询

 电话咨询
电话咨询
联系电话:191 2100 2160
 顶部
顶部
百沐生物孵化于复旦大学,核心团队由复旦大学、中科院、协和医院等知名科研院所的卓越科学家以及来自阿里、美团、小米、生物医药集团等头部互联网科技公司和大型药企集团的综合背景精英组成,专注于将人工智能(AI)技术与生物技术(BT)深度融合,为生命科学研究和精准医疗领域带来创新解决方案。 公司核心平台为自研单细胞平台(BMBIOS)和小沐AI平台(AIGOBIO)。

微信联系我们